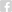Autor: Dinler Amaral Antunes, Bacharel em Biomedicina pela UFRGS, Mestrado e Doutorado pelo PPGBM/UFRGS e pós-doutorado pela Rice University (Texas/EUA).
A minha professora de Bioquímica e tutora na UFRGS, Carmem Gottfried, uma vez nos falou sobre serendipidade na pesquisa. Esta palavra estranha se refere a coisas que acontecem por acaso, oportunidades que surgem, consequências inesperadas de atividades que desempenhamos. E obviamente a ciência está cheia de exemplos de serendipidade. A publicação recente de um artigo nosso na Frontiers in Immunology me fez pensar na inesperada cadeia de eventos que nos trouxe até aqui; na qual o SBlogI desempenhou um papel central.
Em Novembro de 2013 eu participei de um Keystone Symposium on Vaccines, realizado no Rio de Janeiro, e meu trabalho chamou a atenção do Dr. João Carmo. Poucos meses depois eu publiquei um artigo com dinâmica molecular da protease do HIV-1, e recebi o convite do Dr. João Carmo para escrever sobre o trabalho no SBlogI. Este foi o meu primeiro post no blog, e nas postagens seguintes eu resolvi abordar alguns temas de imunoinformática que eram mais relacionados a minha pesquisa de pós-graduação (I, II e III). Não havia nenhuma estratégia de longo prazo envolvida na escolha destes tópicos. Apenas o desejo de compartilhar alguns dos problemas de pesquisa que me interessavam, envolvendo a análise estrutural de complexos peptídeo-HLA. No final de 2014 eu iniciei um pós-doutorado na Rice University nos Estados Unidos, mas continuei trabalhando com modelagem de complexos peptídeo-HLA e continuei contribuindo com o SBlogI.
Algum tempo depois os meus posts chamaram a atenção de uma aluna de Doutorado da USP de Ribeirão Preto, que estava estudando a estrutura e a dinâmica de moléculas de HLA-G. Esta aluna, Thaís Arns, estava sendo orientada pelos professores Eduardo Donadi e Silvana Giuliatti, e ficou empolgada de ver outro pesquisador Brasileiro trabalhando com modelagem de HLAs. Ela seguiu acompanhando as minhas postagens e mais tarde resolveu fazer contato; o que abriu caminho para dois estágios de curta duração em que ela trabalhou comigo no Departamento de Computação da Rice University, sob orientação da professora Lydia Kavraki.
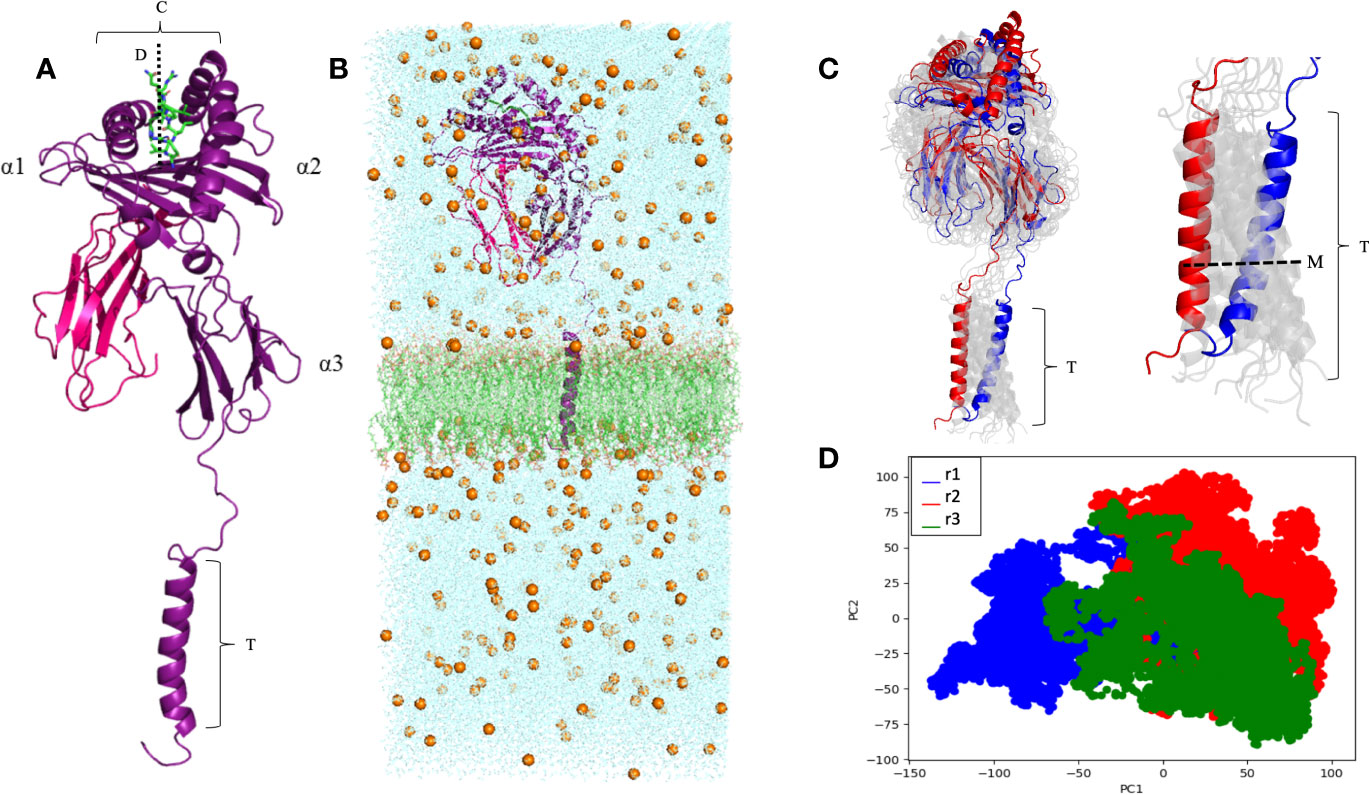
O trabalho da Thaís envolvia inicialmente a modelagem e simulação computacional da molécula completa do HLA-G1 transmembrana (Figura 1), e também do dímero solúvel de HLA-G1. Embora as estruturas do monômero e do dímero do HLA-G1 já haviam sido descritas, estas estruturas estavam incompletas. Além disso, nada se sabia sobre a dinâmica destas moléculas. Por exemplo, foi descrito que o dímero de membrana do HLA-G1 formava um ângulo de 45 graus (Figura 2B), o qual facilitava o acesso de regiões do domínio alfa-3 para a interação com receptores de leucócitos, incluindo receptores ILT4 em células Natural Killer (NK). Mas seria este ângulo imposto pela forma como os protômeros (partes do dímero) estão ancorados na membrana e ligados por uma ponte dissulfeto? Ou seria esta uma conformação estável que se manteria também no dímero solúvel?
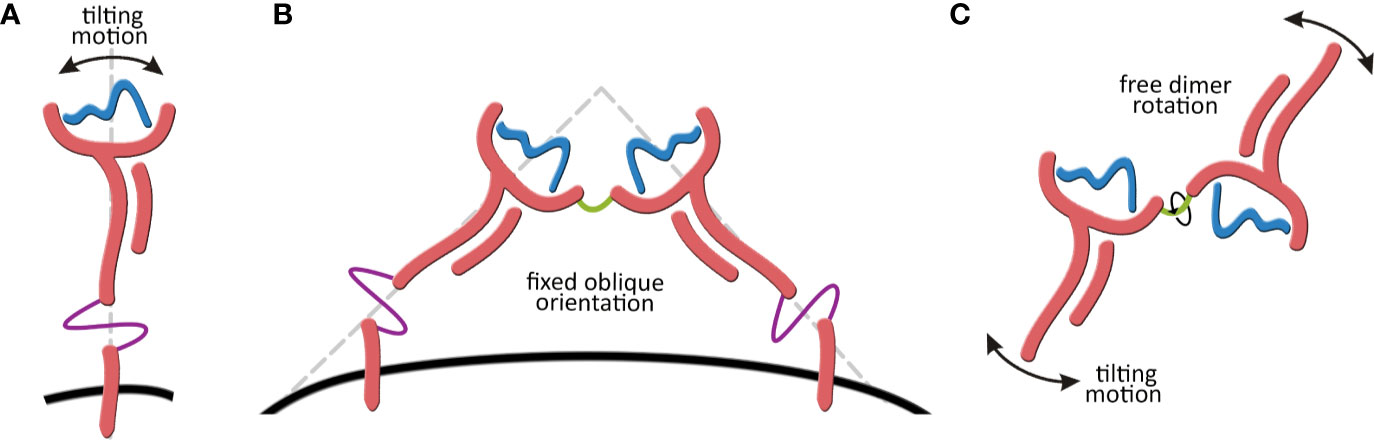
As nossas simulações com o dímero solúvel mostraram que os protômeros são na verdade completamente livres para rotacionar um em relação ao outro (Figura 2C), o que oferece oportunidades completamente diferentes de interação com receptores na superfície de leucócitos. Por exemplo, o modelo de interação entre HLA-G1 e ILT4 proposto em estudos anteriores mostra o ILT4 “se aproximando” pelo lado oposto a membrana (Figura 3A). No entanto, na ausência da membrana e considerando a flexibilidade rotacional do dímero, uma análise de docking proteína-proteína utilizando o servidor ClusPro identificou um modo de ligação alternativo, em que ILT4 “se aproxima” pela base do domínio alfa-3 (Figura 3C).

Além de abrir caminho para novas hipóteses sobre formas de interação do HLA-G com receptores de leucócitos, as quais podem estar diretamente envolvidas com os nuances do papel regulatório das moléculas de HLA-G, este estudo disponibiliza as estruturas 3D geradas (github.com/KavrakiLab/hla-g-models/). Estas estruturas, mais completas do que os cristais disponíveis até então, podem ser utilizadas para futuros estudos estruturais focados na análise de polimorfismos, modelagem e dinâmica de outras isoformas, e interação com outros ligantes de interesse.
Este estudo também estabeleceu uma parceria internacional que continua ativa, envolvendo pesquisadores da USP, da Rice University e da Universidade de Luxemburgo. Após a defesa de seu Doutorado, a Dra. Thaís Arns foi para a Europa (Figura 4), onde hoje desenvolve seu trabalho de pós-doutorado no centro de pesquisa LCSB - Luxembourg Centre for Systems Biomedicine, em Esch-sur-Alzette, Luxemburgo. Seu atual foco de estudo é a proteína DJ-1, alvo de diversas mutações na doença de Parkinson, sob orientação do professor Alexander Skupin. A conexão entre os nossos projetos, materializada agora na forma desta publicação, talvez nunca tivesse ocorrido se não fossem aquelas despretensiosas publicações sobre imunoinformática estrutural no SBlogI. A ciência é cheia de serendipidades, e a comunicação entre pesquisadores e estudantes, por todos os canais possíveis, é essencial para fomentar estes fortuitos e inesperados desfechos.

- Apaixonados por Imunologia
- Comunicado
- Conteúdo Publicitário
- Curso
- Dept. Imunologia Clínica
- Dia da Imunologia
- Dia Internacional da Imunologia
- Divulgação científica
- Edital
- Especial
- Especial Dia da Imunologia
- Especial Doença de Chagas
- Evento
- Eventos
- Exposição COVID-19 da SBI
- História da Imunologia no Brasil
- Homenagem
- Immuno 2018
- Immuno2019
- Immuno2021
- Immuno2022
- Immuno2023
- Immuno2025
- Immuno2026
- IMMUNOLAC
- Immunometabolism2022
- Imune
- Imune - o podcast da SBI
- ImunoWebinar
- INCT Imuno
- Institucional
- IUIS
- Luto
- NeuroImmunology 2024
- Nota
- Nota Técnica
- Notícia
- o podcast da SBI
- Oportunidades
- Outros
- Parecer Científico
- Pesquisa
- Pint of Science 2019
- Pint of Science 2020
- Política Científica
- Sars-CoV-2
- SBI 50 ANOS
- SBI.ImunoTalks
- Sem categoria
- Simpósio
- SNCT 2020
- SNCT 2022
- Webinar
- WORKSHOP
.png)