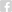Por: Cristina Bonorino* e Helder Nakaya**
Imunologistas são como computadores. A partir do input dos dados obtidos em experimentos, seus cérebros (os “computadores”) irão processar essa informação, cruzando com tudo o que foi aprendido em livros, aulas, artigos, e palestras produzindo, assim, um output que permite responder à pergunta que originalmente provocou esses experimentos. É difícil fazer isso; imunologia é uma ciência complexa. As variáveis parecem ser infinitas. Hoje sabemos que o sistema imune se conecta com praticamente todos os demais sistemas do corpo, influenciando como nos sentimos, ou quanta gordura acumulamos. É também impossível estudar imunologia sem dominar bioquímica, genética, biologia molecular, biofísica, fisiologia, anatomia. Com toda essa complexidade, até recentemente, era muito raro analisar os dados e conseguir produzir previsões. Por exemplo, como aconteceu com o segundo sinal. A sinapse imune entre células T (com seu TCR) e apresentadoras de antígeno (com seu MHC) havia sido visualizada, nos primórdios da microscopia confocal. Mas, enquanto algumas apresentadoras desencadeavam a proliferação das células T após a sinapse, outras não conseguiam fazê-lo. Foi então que, como astrônomos predizem o local exato de um planeta onde nada pode ainda ser observado, diferentes imunologistas predisseram a existência de um segundo sinal, presente apenas em algumas células, que era suficiente e necessário para que a proliferação fosse acionada. Em menos de cinco anos, após inúmeros experimentos em dezenas de laboratórios, o sinal foi identificado: era a proteína B7, hoje conhecida como CD80/CD86.
Isso aconteceu nos anos 1990, quando a internet engatinhava, não existiam smartphones, e o genoma humano ainda não havia sido sequenciado. Nos trinta anos que se seguiram, essas três revoluções influenciaram definitivamente a maneira como os imunologistas trabalham hoje. Ingressamos na era ômica: não apenas genomas, mas transcriptomas, proteomas, metabolomas, lipidomas, epigenomas e outros “omas” são hoje analisados com facilidade, gerando uma quantidade esmagadora de dados. Tão grande, que não podemos armazenar isso em um único computador. Os dados são abrigados em grandes bancos online, podendo ser acessados e analisados com facilidade, inclusive, via smartphone. Os imunologistas, treinados em tecnologias de biologia celular e molecular, vêm-se à frente a de um dilema: a quantidade de dados, pela primeira vez, é infinitamente maior que a capacidade de um humano, ou um time de humanos, de analisá-los. São necessárias ferramentas de inteligência artificial: não apenas computadores, mas mecanismos de busca de dados, seleção de conteúdos, análise e identificação de padrões. Hoje, com o input de dados de dezenas de milhares de estudos em, por exemplo, RNAs expressos em tumores de mama, podemos identificar quais as mutações mais frequentes dentro desses tumores, e que podem predizer um prognóstico bom ou ruim para o paciente, ou a escolha de um coquetel de medicamentos. Atualmente existem diversas opções para o tratamento de câncer; contudo, o mais crítico ainda é poder prever quem vai responder melhor a qual desses tratamentos, otimizando a terapia. A partir dos transcriptomas (ou seja, a análise de expressão dos mais de 25 mil genes humanos) de quem morreu ou sobreviveu à febre amarela, podemos tentar prever a probabilidade de sobrevivência à doença de um paciente assim que ele é internado no hospital. Partindo de dados de transcriptomas e de sintomas apresentados por diferentes indivíduos infectados com vírus Chikungunya, podemos fazer previsões sobre quem vai desenvolver uma artrite de longo prazo ou que logo ficará sem sintomas.
Algumas tecnologias usadas hoje geram, em um único experimento, um nível antes impensável de complexidade e de informação. Por exemplo, imunologistas são hoje capazes de fazer citometria de fluxo com dezenas de marcadores – algo iniciado por uma tecnologia que fundiu Espectrometria de Massas com Citometria – a Citometria de Massas. Um tipo celular é classificado de acordo com a combinação destes marcadores. Uma célula pode tradicionalmente ser chamada de “célula T CD4 humana” se encontrarmos os marcadores CD3 e CD4, mas não encontrarmos os marcadores CD19 e CD8. Se tivermos dezenas de marcadores pra escolher, o número possível de combinações de marcadores será tão grande que apenas a inteligência artificial poderá nos dizer quais utilizar para cada tipo celular. Nesse caso, os algoritmos ainda estão em fase de construção e aperfeiçoamento. Outro exemplo: alguns imunologistas fazem análises clonais de linfócitos, isolando células pela tecnologia de sorting. Porém, hoje é possível medir o transcriptoma dentro de cada célula individual. E foram desenvolvidas novas tecnologias de Single Cell Sequencing que se beneficiam de métodos de aprendizado de máquina para dissecar populações de células heterogêneas sem a necessidade de isolar fisicamente as células por sorting. O uso de inteligência artificial aqui permite a reconstrução das trajetórias de desenvolvimento celular e a modelagem da dinâmica transcricional. Praticamente, uma engenharia reversa de um microambiente complexo.
Os smartphones – que praticamente todas as pessoas possuem hoje – podem ser usados como instrumentos de coletas de dados, como por exemplo, localização. Podemos analisar onde uma pessoa infectada esteve nos últimos dias, e assim identificar possíveis contatos, determinar pontos de transmissão. Nada mais necessário durante a pandemia, possibilitando identificar que ambientes foram ou não visitados por pessoas durante períodos de isolamento – e dessa maneira, prever como a infecção pode se espalhar pela população. Enquanto alguns dos algoritmos que fazem essas análises são privados, muitos são de uso livre e podem ser acessados por quem tem conhecimento de computação.
Esse é um dos gargalos que atualmente limita o uso de inteligência artificial para os imunologistas. Por isso, é tão necessário instituir o treinamento dessas habilidades em todos os programas de imunologia – ou mesmo, já durante a Universidade. Você pode optar hoje por adquirir uma peça de engenharia que usa inteligência artificial e produz previsões, liberando seu tempo para outras atividades no laboratório. Mas, não é má ideia investir um tempo em se familiarizar com essa linguagem. Alguns problemas são tão complexos, que dificilmente um especialista em computação, sozinho, pode resolver sem ter conhecimento profundo de imunologia. Hoje podemos, por exemplo, obter facilmente as sequencias de TCRs em uma população celular, e entender sua composição clonal. Podemos também facilmente identificar os alelos de MHC dos indivíduos, e todas as combinações de peptídeos do proteoma do mesmo microambiente. Podemos inclusive prever, cada vez melhor, que peptídeos podem ser apresentados, com alta probabilidade, por aqueles alelos de MHC em particular. Mas, não sabemos ainda prever que TCRs reconhecem qual combinação MHC-peptídeo. São previsões que requerem uso de múltiplas tecnologias de computação, incluindo analises de bioquímica estrutural. A resolução desse problema revolucionará profundamente não apenas a imunologia básica, mas o desenho de medicamentos e vacinas.
Computadores serão tão bons para a imunologia quanto os imunologistas que os programarem. A qualidade das previsões imunológicas é dependente da qualidade dos dados que disponibilizamos para os processos de aprendizado de máquina. São de silicone os cristais da bola que nos permitirá consolidar a imunologia como uma ciência preditiva.
*Cristina Bonorino é professora titular da Universidade Federal de Ciências da Saúde de Porto Alegre (UFCSPA) e membro do Comitê Científico da SBI Imuno.
** Helder Nakaya é docente da Faculdade de Ciências Farmacêuticas da Universidade de São Paulo (FCF-USP) e membro do Comitê Científico da SBI Imuno.
*** Os conteúdos publicados no SBlogI são autorais e de responsabilidade dos(as) autores(as), não representando a opinião de qualquer patrocinador da Sociedade Brasileira de Imunologia.

- Apaixonados por Imunologia
- Comunicado
- Conteúdo Publicitário
- Curso
- Dept. Imunologia Clínica
- Dia da Imunologia
- Dia Internacional da Imunologia
- Divulgação científica
- Edital
- Especial
- Especial Dia da Imunologia
- Especial Doença de Chagas
- Evento
- Eventos
- Exposição COVID-19 da SBI
- História da Imunologia no Brasil
- Immuno 2018
- Immuno2019
- Immuno2021
- Immuno2022
- Immuno2023
- Immuno2025
- IMMUNOLAC
- Immunometabolism2022
- Imune
- Imune - o podcast da SBI
- ImunoWebinar
- Institucional
- IUIS
- Luto
- NeuroImmunology 2024
- Nota
- Nota Técnica
- Notícia
- o podcast da SBI
- Oportunidades
- Outros
- Parecer Científico
- Pesquisa
- Pint of Science 2019
- Pint of Science 2020
- Política Científica
- Sars-CoV-2
- SBI 50 ANOS
- SBI.ImunoTalks
- Sem categoria
- Simpósio
- SNCT 2020
- SNCT 2022
- Webinar
- WORKSHOP
.png)